Секвенування ДНК
Клонований або ампліфікований фрагмент ДНК можна дослідити різними способами, однак найвичерпнішу інформацію дає встановлення нуклеотидної послідовності (sequence) фрагмента – секвенування. На рис. 8 показано схему найбільш популярного сьогодні методу Сангера (Frederick Sanger). До одноланцюгової ДНК-матриці додається радіоактивно-мічений праймер, повний набір дезоксинуклеозидтрифосфатів (dNTP), ДНК-полімераза та невелика кількість дидезоксинуклеозидтрифосфату одного із чотирьох типів (наприклад, ddATP). Дидезоксинуклеотид відрізняється тим, що містить атом Н замість ОН-групи не тільки при 2′, а також і при 3′ атомі пентози. Включення такого нуклеотиду в ланцюг, що синтезується, приведе до зупинки подальшого зростання ланцюга внаслідок відсутності 3′ ОН-групи на його кінці. Оскільки ddATP присутній у невеликій кількості, така подія буде відбуватися в різних точках ланцюга – в усіх, де аденін розташований напроти тиміну в складі матриці. Шляхом денатурації продуктів реакції отримаємо набір мічених одноланцюгових фрагментів від праймера до кінцевого аденіну. Довжина цих фрагментів у нуклеотидах визначить порядковий номер аденіну в складі ланцюга.
З метою визначення довжини фрагментів проводять гель-електрофорез у денатуруючих умовах, на сусідні лунки гелю наносять також продукти синтезу у присутності інших дидезоксинуклеотидів. Як показано на рис. 8, після електрофорезу та візуалізації смуг із такого гелю можна прочитати нуклеотидну послідовність.
В іншому методі секвенування – методі Максама-Гілберта (Allan M. Maxam, Walter Gilbert) замість ферментативного синтезу застосовують хімічне розрізання ланцюга на нуклеотиді певного типу. Отримані фрагменти різної довжини так само розділяють за допомогою електрофорезу.
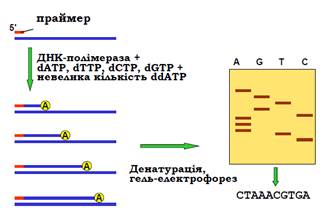
Рис. 8. Секвенування ДНК за Сенгером: схема синтезу ДНК у присутності дидезокси-АТР (ліворуч). Шляхом аналогічної процедури для інших трьох дидезоксинуклеотидів отримуємо набір одноланцюгових фрагментів, що аналізуються за допомогою гель-електрофорезу в денатуруючих умовах (праворуч) – розподіл смуг дозволяє прочитати послідовність (праворуч унизу).
Метод Сенгера використовується сьогодні автоматичними секвенаторами, в яких замість радіоактивно-мічених застосовуються флуоресцентно-мічені праймери. Для кожної з чотирьох реакцій беруть чотири різні флуоресцентні мітки, котрі випромінюють світло в різних спектральних діапазонах. Продукти всіх чотирьох реакцій наносять на гель для електрофорезу разом. Сканування гелю після електрофорезу лазерним променем, що збуджує флуоресценцію, дозволяє дискримінувати продукти різних реакцій, тобто різні кінцеві нуклеотиди, і, таким чином, негайно прочитати послідовність.
Здійснення секвенування кожного із фрагментів, які містяться в геномній бібліотеці клонів, і порівняння послідовностей фрагментів, що перекриваються (рис. 5), дозволяє розмістити клоновані фрагменти в порядку їхньої локалізації в геномі, тобто встановити повну послідовність геному.
Інший сучасний підхід у секвенуванні геномів (так зване піросеквенування), який реалізується на автоматизованих секвенаторах, дозволяє визначити послідовність значно швидше, дешевше і при цьому не потребує ані клонування ДНК, ані електрофорезу. Одноланцюгові фрагменти, отримані з невеликої кількості геномної ДНК, пришивають 5′-кінцями до мікрокульок (один фрагмент на кульку) та ампліфікують за допомогою ПЛР. Кожну кульку із пришитими до неї ампліфікованими ідентичними фрагментами розміщують у мікрореакторі, де здійснюється ДНК-полімеразна реакція. Нуклеозидтрифосфати подаються в реакційну суміш імпульсно один за одним. Якщо нуклеотид певного типу виявляється комплементарним матриці та включається у зростаючий ланцюг, пірофосфат, що при цьому звільняється, залучається до низки хімічних реакцій, де остання реакція супроводжується випромінюванням світла (хемілюмінесценція). Світловий сигнал фіксується оптичною системою, і послідовність таких сигналів читається як нуклеотидна послідовність. Реакція здійснюється паралельно у 200 тис. Мікрореакторів (для 200 тис. фрагментів, які перекриваються), що дозволяє встановити послідовність приблизно 200 млн пар основ за 4,5 години.
Останнім часом з’явилася нова перспектива секвенування ДНК у процесі протягування молекули через нанопору. Спеціалісти із Університету Бостона розробили метод підготовки зразка ДНК до секвенування геному, що є значно швидшим і точнішим від існуючих. Окрім того новий метод дозволяє значно зменшити кількість ДНК, необхідну для аналізу і позбутися дорогого і тривалого етапу ампліфікації ДНК.
Дослідницька група Хаміта Меллера повідомляє про революційну роботу по детекції молекул ДНК, що проходять через нанопори в кремнії. Для продавлювання довгих ниток ДНК через пори (ширина яких становить до 4 нм) дослідники використовують електричне поле. Вимірювання дозволяють фіксувати проходження через нанопору поодинокої молекули нуклеїнової кислоти.
Результати дослідження демонструють можливість визначення і аналізу менших кількостей ДНК, ніж це було можливо до даного часу. Причому, секвенування або профілювання геному з допомогою нанопор дозволить зменшити кількість ДНК, необхідну для аналізу.
У даний час перед секвенуванням ДНК необхідно провести ампліфікацію нуклеїнової кислоти для отримання мільярдів копій, необхідних для процедури розшифрування послідовності пар азотистих основ. Окрім того, що для ампліфікації потрібні додаткові час і ресурси, процес ампліфікації може призводити до спотворення структури продуктів копіювання нуклеїнової кислоти.
Таким чином, було вирішено використовувати електричні поля, локалізовані біля "вихідних отворів" нанопор для впливу на довгі негативно заряджені нитки ДНК і протягування їх через нанопори, після проходження яких молекула ДНК може бути вивчена детальніше. Такий підхід дозволяє використовувати меншу кількість копій ДНК для подальшого аналізу.
Для розробки нового методу дослідники вивчили електрофізичні явища, що проходять на нанорівні, на якому неможливо повноцінно використовувати принципи макроскопічної фізики. У процесі дослідження було виявлено, що нитки ДНК із великою довжиною проходять через пори із більшою швидкістю. Ця обставина дозволяє говорити про те, що система із нанопор може бути оптимізована для детекції довгих ниток ДНК, що складаються із десятків тисяч і навіть більшої кількості пар азотистих основ. Ця обставина може призвести до істотного прискорення процедури аналізу геному, дозволяючи безпосередньо аналізувати довгі ланцюги ДНК, замість того щоб нарізати їх і відновлювати структуру за фрагментами коротких відрізків.
Оскільки існуючі технології ампліфікації ДНК обмежують розміри аналізованої ДНК тисячою парою основ, то новий метод дозволяє позбутися від процедури ампліфікації, він не лише знижує вартість, часові затрати і рівень помилок, характерні для аналізу ДНК попередніми методами, але й дозволяє вивчати нитки ДНК, значно довші, ніж це можливо внаслідок обмежень, характерних для існуючих методів.
Дослідники не зупинилися на досягнутому, продовживши модифікувати розроблену методику. Для зміни параметрів електричного поля біля пор вони застосували градієнти концентрації солей, які дозволили прискорити час захоплення молекул ДНК полем, таким чином, сприяючи швидшому їх проходженню через нанопори і зменшенню кількості ДНК, необхідних для точних вимірювань.
Дата добавления: 2015-09-11; просмотров: 4949;
