Кодоны 507 508 509 510 511 512 513 514 515 516 517 518 519 520 521 522 523 524 525 526 527 528 529
H37RVGGC ACC AGC CAG CTG AGC CAA TTC ATG GAC CAG AAC AAC CCG CTG TCG GGG TTG ACC CAC AAG CGC CGA
455GGC ACG AGC CAG CTG AGC CAA TTC ATG GAC CAG AAC AAC CCG CTG TCG GGG TTG ACC GAC AAG CGC CGA
443GGC ACC AGC GAG CTG AGC CAA TTC ATG GAC CAG AAC AAC CCG CTG TCG GGG TTG ACC GAC AAG CGC CGA
Первичная структура секвенированного rpoB гена у контрольного штамма Mycobacterium tuberculosis H37RV и МБТ, выделенных у больных с множественно резистентным туберклезом. у штамма 455 видны мутации в кодоне 508 и 526, у штамма 443 - в кодоне 510 и 526 rpoB гена. Эти мутации в rpoB гене являются причиной устойчивости возбудителей туберкулеза к рифампицину.
3г. Результатом секвенирования являетсялинейное символьное описание атомной структуры молекулы ДНК, или первичная структура ДНК.
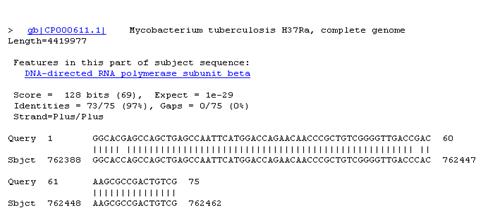
Сравнение секвенированного фрагмента ДНК с международным банком геномов с помощью программы BLAST позволило идентифицировать фрагмент как rpoB Mycobacterium tuberculosis.
4. Первичную структуру секвенированного фрагмента ДНК идентифицируют с использованием программы BLAST, доступ к которой открыт через сервер http://www.ncbi.nlm.nih.gov/blast/.
в) электрофорез в денатурирующем полиакриламидном геле для разделения ампликонов, отличающихся размерами в один нуклеотид;
г) получение линейного символьного описания атомной структуры молекулы ДНК.
4. Биоанализ первичной структуры ДНК с помощью компьютерных программ для получения необходимой информации.
Недостатки секвенирования:
1) позволяет секвенировать фрагменты размером 300 – 1000 нуклеотидов, при этом первые 15–40 и последние 700–900 нуклеотидов распознаются плохо;
2) методы секвенирования ДНК пока недоступны для практических лабораторий, так как требуют дорогостоящего оборудования, дорогостоящих реагентов.
Другие варианты секвенирования.
Крупномасштабное секвенирование, позволяющее секвенировать большие фрагменты ДНК, например, целую хромосому микроорганизмов. ДНК нарезают на фрагменты при помощи рестриктаз. Эти небольшие фрагменты при помощи вектора переносят в Escherichia coli (клонируют). Микроорганизмы, размножаясь приводят к накоплению клонированной ДНК (клональная амплификация). Далее все разновидности клонированных фрагментов ДНК секвенируют и при помощи компьютера собирают в единую длинную цепочку. Такой подход не требует предварительной информации об изучаемой ДНК, поэтому относится к секвенированию de novo.
Секвенирование с использованием гибридизации.В его основу положены гибридизационные биочиповые технологии. При этом неизвестная ДНК обрабатывается флюоресцентным красителем и наносится на чип с большим количеством зондов разной специфичности, находящихся в разных участках чипа. Тот участок чипа, где отмечается сильная флюоресценция, свидетельствует о комплементарности зонда и изучаемой ДНК, что позволяет идентифицировать ДНК, зная характер зонда.
Микрожидкостное секвенирование по Сенджеру. Проводится с использованием всех стадий секвенирования по Сенджеру: ПЦР, очистки продуктов амплификации, электрофореза. Реакция осуществляют в специальных чипах, при этом объем реакции измеряется в нанолитриах, что способствует меньшему расходу дорогостоящих реагентов. Чип состоит из трех функциональных частей: 1) камеры для ПЦР; 2) камеры для очистки ампликонов; 3) камеры для капиллярного электрофореза (капилляр имеет длину 30 см и компактно скручен).
Дата добавления: 2015-02-23; просмотров: 2279;
